Investigadores del IIB-INTECH trabajan en nuevos métodos de diagnóstico para el Chagas. Apuntan al tratamiento temprano de bebés de madres que lo padecen y a encontrar marcadores que indiquen el grado de progreso de la enfermedad.
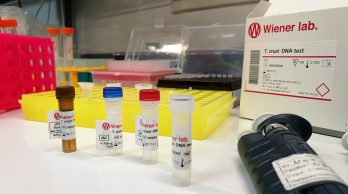
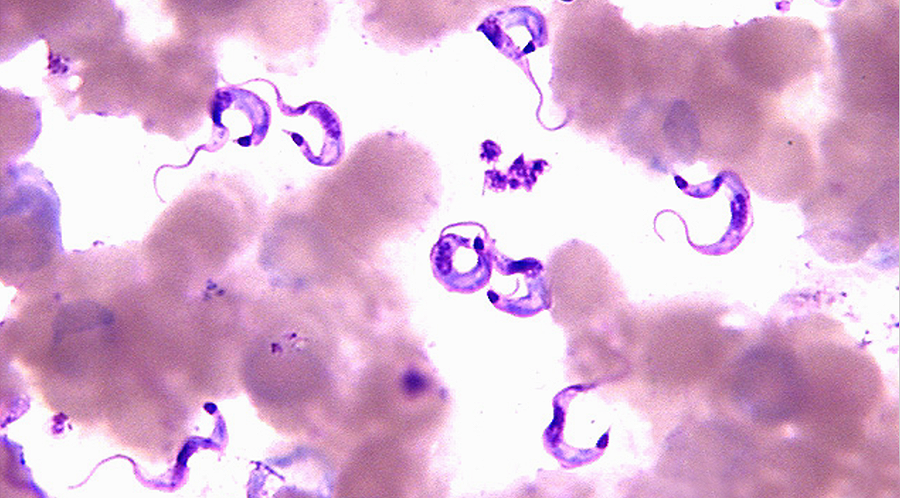
Agencia TSS – A la hora de buscar métodos eficaces de diagnóstico para el Chagas, esta enfermedad suele ser casi tan escurridiza como la vinchuca, el insecto vector del parásito que la transmite (Trypanosoma cruzi). Un método de diagnóstico frecuente para enfermedades infecciosas consiste en tomar una muestra de sangre y observar si el parásito está presente. Es óptimo en caso de que la persona se haya infectado recientemente y esté en lo que se conoce como etapa aguda de la enfermedad, que dura algunos meses. Sin embargo, si la persona ya se encuentra en la etapa posterior, llamada crónica, es difícil hallar parásitos en una muestra de sangre porque son pocos, a pesar de que siguen generando consecuencias negativas en la salud del paciente.
En este caso, el método de diagnóstico se basa en la reacción del sistema inmune. Ante una enfermedad infecciosa, el organismo genera anticuerpos específicos que solo se forman a partir del contacto con el microorganismo. Por lo tanto, si se observa la presencia de esos anticuerpos, significa que la persona está infectada. Como se genera una cantidad muy grande, con una pequeña muestra de sangre es suficiente. Pero aquí aparece un nuevo desafío: es necesario conocer al sistema inmune lo mejor posible para saber cómo reacciona ante determinados antígenos (es decir, cualquier molécula ajena al organismo que desencadena una respuesta inmunitaria).
Tras ese objetivo va el equipo de científicos dirigido por Fernán Agüero (foto), perteneciente al Instituto de Investigaciones Biotecnológicas (IIB -INTECH) de la UNSAM. Agüero es biólogo doctorado en química, especialista en bioinformática y comenzó a investigar la genómica del T. cruzi a fines de los años 90. Así, fue desarrollando una línea de investigación que, a través de sucesivas tesis realizadas por estudiantes e investigadores de la UNSAM derivó, entre otros logros, en el desarrollo de un microchip de péptidos, una plataforma que permite reconocer la presencia de anticuerpos específicos. Gracias a este avance, que posibilita realizar múltiples ensayos simultáneos a partir de pequeñísimos fragmentos de proteínas (los péptidos), el equipo ha conseguido duplicar el número de antígenos conocidos para la enfermedad de Chagas.
«Nuestro trabajo está enfocado en tratar de desarrollar un método de diagnóstico para el Chagas congénito. El problema con los bebés de madres chagásicas es que, al sacar una muestra de sangre, da positivo por más que no estén infectados debido a que reciben la sangre de la madre por el cordón umbilical. Entonces, es necesario desarrollar un método de diagnóstico que permita diferenciar la respuesta inmune del bebé y de la madre. Esto es muy importante porque si el bebé infectado es tratado de manera temprana las chances de éxito son muy altas», explica Agüero.
Una aliada principal en la búsqueda de métodos más eficaces de diagnóstico y tratamiento de enfermedades es la bioinformática. Esta disciplina aprovecha los avances en la biología molecular y en la computación para refinar los análisis de la abundante cantidad de datos obtenidos en laboratorio. «Lo bueno de la informática es que, una vez que se desarrolla una herramienta, por más que esté diseñada para una enfermedad en particular, como el Chagas, es relativamente sencillo adaptarla y aplicarla a otra de características similares», indica el biólogo.
De esta manera, una vez modelado y diseñado un método en la computadora, los investigadores definen qué péptidos van a probar en el microchip a partir de muestras de sangre obtenidas de pacientes chagásicos y de personas sanas. Allí, observan la respuesta del sistema inmune: qué anticuerpos genera y contra qué péptidos. Los resultados obtenidos vuelven a la computadora para continuar refinando el método y pasar al siguiente ensayo.
En busca de una vacuna
Cuando un organismo se enfrenta con un patógeno genera una respuesta inmune, no contra el patógeno de manera global, sino contra determinadas moléculas o partes de ellas. Sin embargo, no es fácil saber cuáles son los blancos «elegidos» por el sistema inmune ni por qué los elige. Esta dificultad varía, además, según la complejidad del microorganismo en cuestión: el genoma de un virus, por caso, se puede descomponer en unos 3.000 péptidos cortos diferentes, mientras que para el del T. cruzi se necesitarían más de 6 millones. Además, cada organismo reacciona de manera diferente, aunque hay ciertos patrones que son universales. De otro modo, no sería posible la elaboración de medicamentos y vacunas.
Hasta hace unas décadas, para determinar si una persona estaba infectada se medía la reacción de una muestra de sangre ante un parásito entero o partes de él. Con el tiempo, la técnica se fue mejorando y se empezaron a utilizar moléculas: genes y proteínas recombinantes. “Hoy podemos sintetizar en un chip más de un millón de ‘pedacitos’ de proteínas (los péptidos) del parásito y ver en qué lugar específico de ese chip hubo reacción. Estamos aprovechando una miniaturización muy grande en la plataforma para poder ver con mucho detalle la infinidad de interacciones que se producen entre el sistema inmune y los diversos antígenos”, señala Agüero.
El sistema inmune funciona a través de dos tipos de respuestas: la parte humoral, compuesta por las células B, que son las que producen anticuerpos; y la parte celular, compuesta por las células T. En la primera, los anticuerpos reconocen determinadas moléculas (esas que eligen como su “blanco”) y se pegan a ellas. Así generan una señal para que el sistema inmune vaya tras ellas. A diferencia de los anticuerpos, la respuesta T es ejercida por células, que atacan al patógeno y a otras células del cuerpo que reconocen como infectadas.
En la búsqueda de una vacuna contra el Chagas, Agüero dice que “una buena solución tendría que estimular las dos partes del sistema inmune”. Actualmente, hay dos drogas aprobadas para el tratamiento contra el Chagas: benznidazol y nifurtimox. “No hubo muchas mejoras en el desarrollo de drogas desde los años 70. Estas dos funcionan, pero, aunque en chicos prácticamente no producen efectos secundarios, tienen muchos efectos colaterales en adultos”, apunta el investigador.
Para Agüero, la principal dificultad para encontrar una vacuna contra el Chagas, más allá de la complejidad del propio parásito, es la falta de inversión. “No es una enfermedad atractiva en lo económico porque el mercado de los que pueden llegar a consumir las drogas o los diagnósticos es de bajos recursos. Entonces, las farmacéuticas prefieren desarrollar drogas para otras enfermedades, como cáncer, diabetes o afecciones cardíacas. Por eso, la inversión para las enfermedades llamadas desatendidas proviene de los sistemas de salud pública, pero aun así no es suficiente”, considera. De todos modos, el especialista indica que, en la última década, las farmacéuticas han invertido más recursos en este tipo de enfermedades.
Los principales objetivos del grupo de investigación son la obtención de un método de diagnóstico para el Chagas congénito y la búsqueda de nuevos marcadores que indiquen el éxito o fracaso de un tratamiento. “Una de las razones por las que no se han desarrollado nuevas drogas contra el Chagas es que los ensayos clínicos en pacientes para testear su efecto suelen ser muy largos. En general, el paciente chagásico empieza a tener complicaciones recién 20 o 30 años después de infectado. Realizar ensayos durante tanto tiempo es muy costoso. Por eso, queremos encontrar marcadores que indiquen si un tratamiento funciona sin necesidad de esperar tanto”, concluye.
![]()
27 sep 2016
Temas: Bioinformática, Chagas, Computación, IIB, Salud, UNSAM