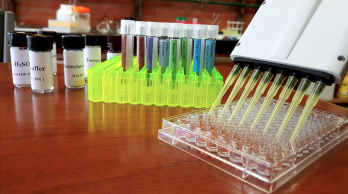
Científicos del CONICET y de la UBA trabajan en el desarrollo de FluoTB, una técnica que usa virus modificados para diagnosticar tuberculosis de forma más rápida, precisa y económica que las técnicas actuales. El desarrollo recibió el premio César Milstein en el área de biotecnología y crearon una empresa para lanzarlo al mercado.
Agencia TSS – La tuberculosis es una enfermedad que provoca diariamente más de 3000 muertes en el mundo y genera cerca de 30.000 nuevos contagios, por lo que lograr diagnósticos rápidos es una parte fundamental del tratamiento. Mariana Piuri, investigadora del CONICET en el Instituto de Química Biológica (IQUIBICEN, CONICET-UBA), desarrolló junto con su equipo una técnica para detectar tuberculosis de forma rápida, precisa y a bajo costo. El método utiliza virus que hacen «brillar» a las bacterias infectadas y fue uno de los ganadores del Premio César Milstein 2025 a la investigación en biotecnología con impacto en salud.
En la Argentina, en 2024, se notificaron alrededor de 16.600 casos de tuberculosis, de los cuales aproximadamente el 84 por ciento correspondieron a personas de 20 a 44 años. Los métodos diagnósticos tradicionales presentan varias limitaciones. Uno de los más utilizados es la baciloscopía, que consiste en tomar una muestra de esputo del paciente, teñirla con una sustancia especial y observarla al microscopio para detectar la bacteria que causa la tuberculosis. Sin embargo, tiene baja sensibilidad, lo que significa que puede no detectar la enfermedad si hay pocas bacterias en la muestra. “Hay muchos casos que se pierden, sobre todo en pacientes que no tienen una carga bacteriana muy alta”, explica Piuri.
Por otro lado, los métodos genotípicos, aunque más rápidos, también enfrentan restricciones, dado que no pueden diferenciar si las bacterias detectadas están vivas o muertas. Durante el tratamiento, se sigue detectando ADN bacteriano incluso cuando las bacterias ya no están activas: “Cuando un paciente empieza a recibir antibióticos, tiene que volver al laboratorio cada uno o dos meses para evaluar si el tratamiento está funcionando realmente”, señala la investigadora.
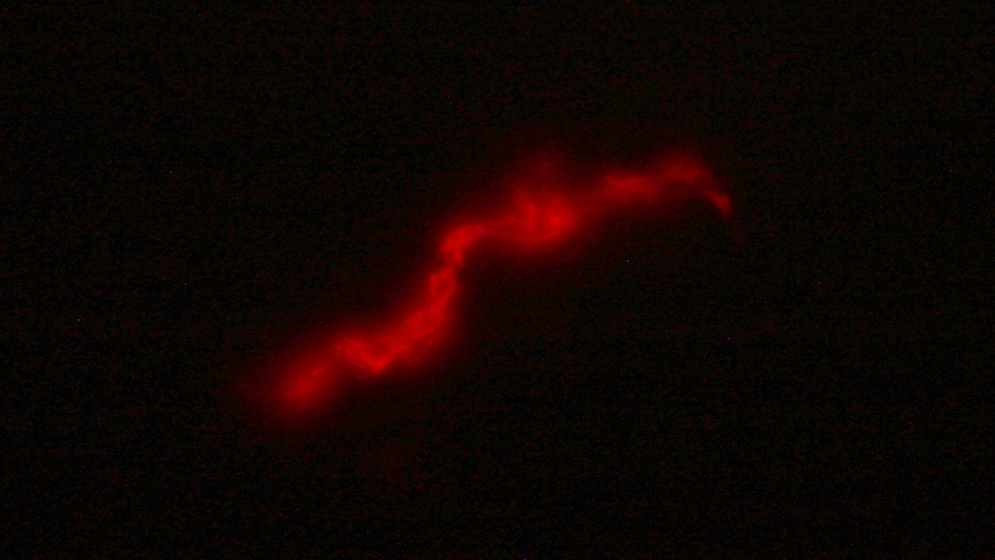
El nuevo método desarrollado por el equipo liderado por Piuri se basa en el uso de bacteriófagos, virus especializados en infectar bacterias, particularmente las micobacterias como la que causa la tuberculosis. Con una modificación genética, lograron convertir estos virus en detectores precisos de la enfermedad: “Modificamos el genoma de este fago (el bacteriófago) e introducimos un gen que codifica un reportero fluorescente”, explica Piuri. “Usamos una versión roja llamada MCherry. Si el fago se encuentra con la bacteria, el reportero se expresa y la bacteria se vuelve roja”, agrega.
El procedimiento es sencillo en comparación con las técnicas tradicionales. Se toma una muestra del esputo del paciente, se la procesa para eliminar otras bacterias que puedan interferir con el análisis, y luego se la pone en contacto con los bacteriófagos modificados.“Si con el microscopio se ve una bacteria roja fluorescente, quiere decir que esa muestra tenía micobacterias. Esto podemos hacerlo en tan solo tres a cinco días desde que se toma la muestra”, aclara la científica. Además de detectar tuberculosis, el sistema permite distinguir entre diferentes tipos de micobacterias. “Tenemos una manera de saber si esa micobacteria presente en la muestra es tuberculosis o es otra especie”, añade.
Práctico y accesible
Una de las ventajas clave del nuevo método es que permite diferenciar bacterias vivas de bacterias muertas, algo que los métodos genotípicos no pueden hacer: “Cualquier técnica que se limite a detectar ADN va a seguir dando positivo aunque las bacterias estén muertas, porque el material genético sigue ahí”, explica Piuri. “Nuestros fagos solo infectan bacterias viables que están vivas. Por eso, nuestra metodología hace una gran diferencia”, dice.
Esto no solo permite confirmar diagnósticos con mayor precisión, sino también evaluar si un tratamiento está funcionando. El sistema incluso permite identificar si las bacterias son resistentes a ciertos antibióticos: “Si una bacteria es resistente, va a seguir viva aunque esté en contacto con la droga, y el virus la va a infectar, encendiendo la señal fluorescente. Si es sensible, la bacteria muere y el fago no puede hacer nada. Así, podemos saber rápidamente si esa cepa es resistente o no”, explica.
Esta información es crucial para frenar la transmisión de cepas resistentes. “Si un paciente no recibe el antibiótico adecuado, sigue contagiando y diseminando bacterias resistentes. Además, el tratamiento puede durar hasta dos años en esos casos, y muchos pacientes lo abandonan, lo que agrava aún más el problema”, señala Piuri. Otra ventaja destacada del nuevo método es su costo potencialmente mucho más bajo:“Hoy un método genotípico en Argentina cuesta alrededor de 100 dólares por muestra. Nuestra metodología, aunque aún no tiene un precio final, está pensada para ser mucho más accesible”, afirma.
Esto tiene especial relevancia en el contexto del sistema de salud argentino. “La mayoría de los diagnósticos de tuberculosis se hacen en hospitales públicos. Son pocos los laboratorios privados que trabajan con esta enfermedad”, explica. Además, el nuevo método no requiere equipamiento costoso ni infraestructura compleja. “Esta herramienta puede utilizarse en laboratorios con microscopios de fluorescencia, que ya existen en la mayoría de los centros donde se hace diagnóstico de tuberculosis”, dice.
Esta simplicidad técnica permitiría descentralizar el diagnóstico, una estrategia clave para llegar a regiones donde la enfermedad tiene mayor incidencia: “En el noroeste del país, por ejemplo, la tasa de tuberculosis es más alta. Este método permitiría testear localmente todas las muestras de pacientes con sospecha y actuar más rápido”, concluye Piuri.
Más de una década de investigación
El desarrollo de esta tecnología llevó más de una década de trabajo y refleja los desafíos de llevar la investigación básica al terreno clínico. Los primeros fagos fueron desarrollados mientras la científica realizaba su posdoctorado en el Pittsburgh Bacteriophage Institute, en la Universidad de Pittsburgh, en Estados Unidos. En 2010, regresó a la Argentina con la intención de continuar y potenciar ese trabajo, ahora desde su propio grupo de investigación: “Cuando ya estaba en el país, con mi equipo empezamos a optimizar esa primera versión de los fagos para mejorarla”, explica.
Así nació el Laboratorio de Bacteriófagos y Aplicaciones Biotecnológicas, que logró diseñar una segunda generación de estos bacteriófagos reporteros, con mejoras en su sensibilidad, estabilidad y potencial diagnóstico. El avance crucial fue la evaluación en muestras reales: “Decidimos que era el momento de evaluarlo en muestras clínicas de pacientes. Hasta ese momento, todo lo que se había hecho con estos fagos había sido en el contexto del laboratorio de investigación. Teníamos que saber qué pasaba cuando lo llevábamos a un entorno real y clínico”, relata la investigadora.
La colaboración con el Laboratorio de Bacteriología de la Tuberculosis del Hospital Muñiz, y con el Instituto Raúl Vaccarezza, fue fundamental. “Tienen mucha experiencia en tuberculosis y se entusiasmaron mucho con el proyecto», señala Piuri. Esta alianza permitió evaluar la tecnología en aproximadamente 300 muestras de pacientes, obteniendo “muy buenos resultados, con altos valores tanto de sensibilidad como de especificidad”.
El siguiente paso era transformar la investigación en un producto de diagnóstico. “Nosotros sabíamos que ya había funcionado con muestras clínicas, pero siempre a mini escala, y había que transformar eso en un kit verdadero”, explica Piuri. “Tenemos que escalar todo esto a un nivel de producción, hay que pensar cómo va a ser el formato del kit”, agrega.
Sin embargo, el recorrido tuvo sus propios obstáculos. “Nos agarró la pandemia en el medio, que nos frenó muchísimo porque no logramos tener los permisos en la Facultad para poder ir a trabajar en este proyecto”, recuerda la científica. Otro inconveniente llegó cuando la primera empresa biotecnológica que había seleccionado el proyecto decidió no continuar. “Nos quedamos desmotivados, en un momento estuve a punto de abandonar el proyecto porque habíamos puesto mucho esfuerzo”, admite Piuri.
Pero desde adentro surgió una nueva idea que se pudo materializar. Fueron dos estudiantes del laboratorio que realizan sus tesis doctorales, Florencia Payaslián y Agustín Seijo, quienes propusieron una nueva estrategia: “Pensaron en avanzar por otro camino y la idea fue armar una start up, de la que los tres somos los cofundadores”, dice.
La científica reconoce la importancia de los socios comerciales en este proceso: “Es muy difícil que podamos hacerlo solos, no por una cuestión solo de recursos, que obviamente se necesitan, sino también por una cuestión de expertise que como científicos no tenemos. Entonces, ahí es donde hay que recurrir a socios comerciales y a gente que conoce de otros temas”.
El potencial del proyecto trasciende las fronteras nacionales. “En cuanto a la posibilidad de internacionalizar el proyecto, cuando vos entrás en este mundo de las start ups, los inversores ya no solo piensan en el mercado local, sino que obviamente apuntan a otros mercados”, explica Piuri.
Aún así, el contexto actual presenta dificultades para la continuidad de la investigación. “Lamentablemente, en este momento el financiamiento que tenemos es nulo. Nosotros todos estos años siempre tuvimos financiamiento de diversos tipos», señala la investigadora. “Siempre el financiamiento que hacía la diferencia era de la Agencia (I+D+i), eso permitió que pudiéramos avanzar con este proyecto”.
La reducción drástica de los fondos públicos obligó a los científicos a buscar alternativas. “Al desaparecer el financiamiento de la Agencia, sumado a otros que se cortaron prácticamente en su totalidad, la idea de presentarnos a este premio tuvo que ver también con que estamos todos los investigadores buscando fuentes alternativas de financiamiento”, explica Piuri, refiriéndose al premio César Milstein 2025.
Un dato revelador fue que a este premio se presentaron 78 proyectos, 50% más de lo que se presentaban normalmente. “Eso muestra que todos los investigadores estamos tratando de buscar fuentes de financiamiento alternativo para poder seguir avanzando con los proyectos, porque en este momento el aporte que tenemos es prácticamente nulo”.
A pesar de la situación del país, el proyecto sigue adelante: “Ahora estamos en un período de incubación y esperando la respuesta final, si se va a hacer la inversión o no. En base a eso va a depender de cuánto va a tardar en salir FluoTB al mercado. Pero confiamos en que va a ser en poco tiempo”, concluye la investigadora.
![]()
08 ago 2025
Temas: CONICET, Salud, Start ups, Transferencia, Tuberculosis